Aplogruppi del cromosoma Y
Da Ufopedia.
(→Aplogruppi A e B) |
(→Aplogruppi CT (DE ed C,F)) |
||
| Riga 80: | Riga 80: | ||
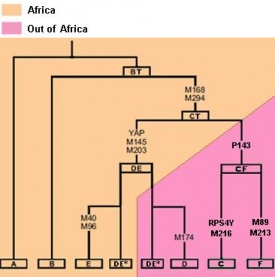
Le mutazioni M168 e M294 (CT o CR), assenti in A e B, definiscono tutti gli aplogruppi da C a T. Queste mutazioni precedettero la migrazione ''fuori dall'Africa'', essendo presenti sia in Africa che al di fuori. Le mutazioni che caratterizzano DE (M145, M203) si verificarono in Africa più di 50.000 anni fa. | Le mutazioni M168 e M294 (CT o CR), assenti in A e B, definiscono tutti gli aplogruppi da C a T. Queste mutazioni precedettero la migrazione ''fuori dall'Africa'', essendo presenti sia in Africa che al di fuori. Le mutazioni che caratterizzano DE (M145, M203) si verificarono in Africa più di 50.000 anni fa. | ||
L'aplogruppo E rimase originariamente in Africa e le sue più alte frequenze si riscontrano in Africa Sub-Sahariana occidentale (81%) e Etiopia (68%). Il sub-clade E1b è di origine africana e si disperse per tutto il mediterraneo raggiungendo la frequenza del 27% in Grecia. | L'aplogruppo E rimase originariamente in Africa e le sue più alte frequenze si riscontrano in Africa Sub-Sahariana occidentale (81%) e Etiopia (68%). Il sub-clade E1b è di origine africana e si disperse per tutto il mediterraneo raggiungendo la frequenza del 27% in Grecia. | ||
| - | [[Immagine: | + | [[Immagine:CM130-Migration.jpg|thumb|350px|Il percorso della migrazione di M130, la mutazione che caratterizza l'aplogruppo C]] |
L'aplogruppo D si trova soltanto in Asia, soprattutto nell'Himalaya e in Giappone, dove fu introdotto dai primi colonizzatori. | L'aplogruppo D si trova soltanto in Asia, soprattutto nell'Himalaya e in Giappone, dove fu introdotto dai primi colonizzatori. | ||
Le mutazioni tipiche che definiscono gli aplogruppi citati nel testo: | Le mutazioni tipiche che definiscono gli aplogruppi citati nel testo: | ||
Versione delle 08:57, 7 giu 2011
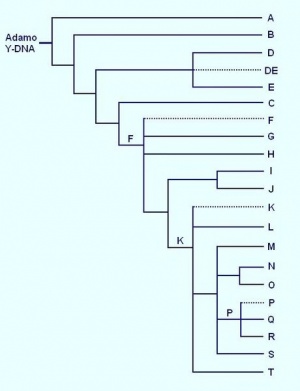
In genetica umana, gli aplogruppi del cromosoma Y sono raggruppamenti di combinazioni di marcatori (aplotipi) definiti dalle differenze nella regione non-ricombinante del DNA del cromosoma Y (chiamato NRY da Non-Recombining Y-chromosome). Queste differenze fanno riferimento a polimorfismi biallelici (SNPs, Single Nucleotide Polymorphisms).
IL YCC Y Chromosome Consortium ha stabilito un sistema per definire gli aplogruppi del cromosoma Y basato sulle lettere da A a T, con ulteriori divisioni usando numeri e lettere in pedice.
Il cromosoma Y ancestrale (scherzosamente definito dagli studiosi di "Adamo") è quello appartenuto ad un maschio teorico che rappresenta il più recente progenitore comune (MRCA Most Recent Common Ancestor) di tutti i maschi attuali lungo la linea patrilineare, visto che il cromosoma Y è unicamente trasmesso dal padre ai figli maschi.
La stima di quando questo individuo teorico sia vissuto varia a seconda degli studi.
Indice |
Albero filogenetico dell'umanità (cromosoma Y)
Y, il cosiddetto "'Adamo ancestrale'"
- A (M91, P97): Nell'Africa subsahariana, specialmente tra i Boscimani (dell'etnia Khoisan)[1] e Sudanesi.
- BT (SRY1532.1/SRY10831.1, M42, M94, M139, M299) (Questo gruppo è indicato anche come YxA.)
- B (M60, M181, P85, P90): Diffuso in gran parte dell'Africa subsahariana, particolarmente tra i Pigmei[1] e gli Hadza.
- CF o CT (M168, M294, P9.1)
- DE (M1/YAP, M145/P205, M203, P144, P153, P165, P167, P183)
- DE*: Poco nella Nigeria.[2]
- D (M174, 021355): Proprio dell'Asia orientale,[3] specialmente nel Giappone, Tibet e isole Andamane.
- E (M40/SRY4064/SRY8299, M96, P29, P150, P152, P154, P155, P156, P162, P168, P169, P170, P171, P172, P173, P174, P175, P176): Copre la parte più vasta dell'Africa. Presente anche nel Vicino Oriente e nell'Europa meridionale.[4]
- C,F (P143)
- C (M130/RPS4Y711, M216, P184, P255, P260): Nell'Eurasia orientale.
- C1: Poco nel Giappone.[5]
- C2: Nelle isole del Pacifico che comprende la Polinesia, la Micronesia e la Melanesia.
- C3: In una vasta area geografica compresa fra l'Asia centrale, l'Asia orientale, la Siberia e fra i Nativi americani dell'America del Nord.
- C4: Un'alta frequenza si ha negli Australiani aborigeni.[6]
- C5: Nell'Asia meridionale. Anche nell'Asia centrale e nel Vicino Oriente.
- C6: Nella Nuova Guinea.[7]
- F (M89, M213/P137, M235, P14, P133, P134, P135, P136, P138, P139, P140, P141, P142, P145, P146, P148, P149, P151, P157, P158, P159, P160, P161, P163, P166, P187)
- F*: Specialmente negli popoli tribali indigeni dell'India.[8]
- G (M201, P257, U2, U3, U6, U7, U12, U17, U20, U21, U27, U33): Le maggiori frequenze si riscontrano nel Caucaso.[9] Presente anche tra i Mediorientali e nell'Europa meridionale.
- H (M69, M370): Principalmente attraverso il Subcontinente indiano[10] e negli zingari.
- IJK (L15/M523/S137, L16/M522S138, L69.1(=G)/S163.1)
- IJ (M429/P125, P123, P124, P126, P127, P129, P130, S2, S22)
- K (M9, P128, P131, P132)
- K*: Specialmente nell'Oceania.
- LT (L298/P326)
- L (M11, M20, M22, M61, M185, M295): Principalmente nell'Asia meridionale.[13]
- T (M70, M184/USP9Y+3178, M193, M272) Diffuso nell'Europa, nel Vicino Oriente, nell'India, nel Corno d'Africa e altre regioni.
- MNOPS (M526)
- M (P256): Prevalente nella Melanesia.[14]
- NO (M214, P188, P192, P193, P194, P195)
- N (M231): Fino all'Estremo Oriente, la Siberia e comune tra i popoli uralici.[15]
- O (M175, P186, P191, P196): Prevalente nell'Asia orientale e nel Sud-est asiatico, con una frequenza prossima al 75%.[16]
- P (92R7, M45, M74/N12, P27.1/P207, P69, P226, P228, P230, P235, P237, P239, P240, P243, P244, P281, P282, P283, P284, P295)
- Q (M242): È l'aplogruppo principale in quasi tutti i Nativi americani.[17]
- R (M207/UTY2, M306/S1, P224, P227, P229, P232, P280, P285, S4, S8, S9, V45)
- R1 (M173)
- R2 (M124): Importante nel Subcontinente indiano.[20]
- S (M230,P202, P204): Principalmente nella Papua Nuova Guinea.[14]
- C (M130/RPS4Y711, M216, P184, P255, P260): Nell'Eurasia orientale.
- DE (M1/YAP, M145/P205, M203, P144, P153, P165, P167, P183)
Migrazioni
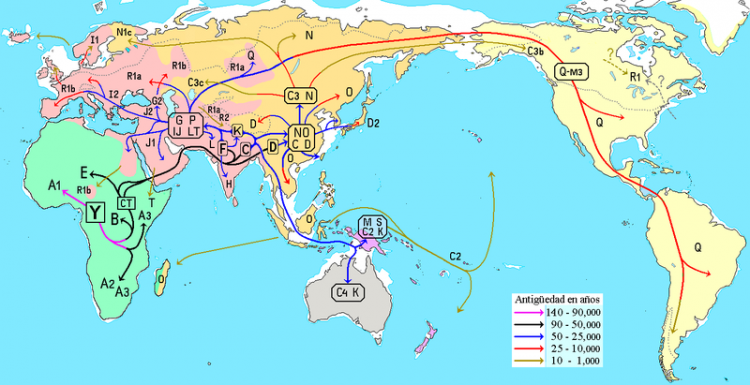
Gli umani moderni secondo studi del cromosoma Y è originario dell'Africa subsahariana e ha poi colonizzato l'Eurasia (circa 70 mila anni fa)[21] seguendo la costa meridionale dell'Asia.[22] I gruppi che partono dall'Africa, si sarebbero successivamente dispersi secondo il seguente mappa:
Aplogruppi principali
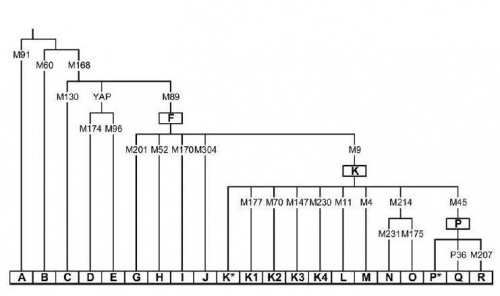
I principali aplogruppi del cromosoma Y comprendono:
Aplogruppi A e B
In nessuna parte del mondo sono stati trovati lignaggi del cromosoma Y più antichi di 90.000 anni. Gli aplogruppi A e B sono considerati i più antichi e si trovano soltanto nell'Africa Sub-Sahariana o in popolazioni con la stessa origine come, per esempio, gli Afroamericani portati oltreoceano con la tratta degli schiavi. Le frequenze più importanti di A si trovano tra i Boscimani, i Khung e i Sudanesi. Le frequenze più elevate di B tra i Pigmei Biaka e Mbuti. Come si evince dall'albero filogenetico degli aplogruppi del cromosoma Y, tutta la diversità moderna si è generata in Africa, per cui le popolazioni contemporanee sono discendenti degli Africani che restarono in Africa o di quelle popolazioni che emigrarono fuori dall'Africa per popolare il resto dei continenti.
La prima biforcazione dal tipo ancestrale teorico (di Adamo) deriva dalla mutazione M91 che definisce l'aplogruppo A (dal quale derivano A1, A2 e A3). Tutti gli altri aplogruppi derivano da BT (o BR noto anche come YxA, che significa: tutti i cromosomi Y che non appartengono all'aplogruppo A). Nell'elenco che segue sono indicate le mutazioni che caratterizzano gli aplogruppi menzionati in questa sezione:
- A-M91
- A1-M31
- A2-M6
- A3-M32
- BT (o BR) M42, M94, M139, M299
- B-M60
- B1-M236
- B2-M182
- CT (vedi il seguito)
- B-M60
Aplogruppi CT (DE ed C,F)
Le mutazioni M168 e M294 (CT o CR), assenti in A e B, definiscono tutti gli aplogruppi da C a T. Queste mutazioni precedettero la migrazione fuori dall'Africa, essendo presenti sia in Africa che al di fuori. Le mutazioni che caratterizzano DE (M145, M203) si verificarono in Africa più di 50.000 anni fa. L'aplogruppo E rimase originariamente in Africa e le sue più alte frequenze si riscontrano in Africa Sub-Sahariana occidentale (81%) e Etiopia (68%). Il sub-clade E1b è di origine africana e si disperse per tutto il mediterraneo raggiungendo la frequenza del 27% in Grecia.
L'aplogruppo D si trova soltanto in Asia, soprattutto nell'Himalaya e in Giappone, dove fu introdotto dai primi colonizzatori. Le mutazioni tipiche che definiscono gli aplogruppi citati nel testo:
- CT (M168, M294)
- DE (M1, M145, M203) ca. 50 kya
- D (M174)
- E (M40, M96)
- E1
- E2
- CF (P143)
- C (M130, M216)
- F o GT (P14, M89) (vedi il seguito)
- DE (M1, M145, M203) ca. 50 kya
Aplogruppo F (GT)
Gli aplogruppi che discendono dal'aplogruppo F rappresentano il 90% della popolazione mondiale, ma si distribuiscono quasi esclusivamente fuori dall'Africa sub-sahariana. IJ corrisponde probabilmente a una ondata migratoria dal Medio-Oriente o all'Asia occidentale a partire da 45.000 anni fa, che si è poi diffusa in Europa con l'uomo di Cro-Magnon. L'aplogruppo G, originatosi anch'esso in Medio-Oriente, o nel Caucaso, o forse più a Est in Pakistan, intorno a 30.000 anni fa, secondo alcuni studi potrebbe essersi diffuso in Europa nel Neolitico, oppure, vista la sua forte discontinuità, aver raggiunto l'Europa già nel Paleolitico. L'aplogruppo H si originò forse in India 30-40.000 anni fa, dove persistette fino a ridiffondere in epoche storiche con i Gitani. L'aplogruppo K si originò probabilmente nell'Asia sud-occidentale e da lì si diffuse in Africa, Eurasia, Australia e Sud-Pacifico. Mutazioni che caratterizzano gli aplogruppi citati nella sezione:
Aplogruppo I
L'aplogruppo I rappresenta circa un quinto dei cromosomi Y europei. È quasi esclusivo dell'Europa pertanto si ritiene che si sia originato in quest'area prima dell'ultima glaciazione. È probabile che sia stato confinato nel rifugio balcanico durante la glaciazione e che poi si sia ridiffuso verso nord con il ritiro dei ghiacciai. Nonostante sia relativamente frequente negli Scandinavi, nei Sardi e nelle popolazioni balcaniche, questi popoli presentano subcladi differenti dell'aplogruppo I. Questo suggerisce che ognuna delle popolazioni ancestrali è oggi dominata da un particolare subclade che ha marcato una indipendente espansione della popolazione lungo diversi percorsi migratori durante e immediatamente dopo la glaciazione.
Le principali sub-cladi dell'aplogruppo I sono:
I1 (M253, M307, P30, P40) (ex I1a) con le più alte frequenze in Scandinavia, Islanda, e Europa nord- orientale. Nelle Isole britanniche la mutazione I1-M253 è spesso usata come marcatore delle invasioni vichinghe o anglosassoni.
I2 (S31) (ex I1b), che include I2a (P37.2) (ex I1b1 ed ex I1b) è la forma più comune nei Balcani e in Sardegna (dove rappresenta l'aplogruppo più cospicuo nella variante I2a1 (M26)) (ex I1b1b) e I2b (ex I1b2) che raggiunge discrete frequenze lungo le coste nord-occidentali dell'Europa continentale e in Sardegna. Dalla linea I2b è derivato I2b1a (M284) in Europa nord-occidentale ed Isole Britanniche.
Aplogruppo K (LT)
L'aplogruppo L si trova principalmente nel sud dell'Asia. L'aplogruppo M è prevalente nelle isole Papua e Nuova Guinea. Gli aplogruppi N e O comparvero 35-40.000 anni fa in Asia orientale.
L'aplogruppo N si originò probabilmente in Mongolia e si diffuse fino all'estremo Oriente e la Siberia come ad ovest, essendo il gruppo più comune tra i popoli uralici. L'aplogruppo O si trova in Estremo Oriente. L'aplogruppo P si trova soprattutto come sottotipi Q o R (vedi il seguito), raramente non differenziato. Si evolse probabilmente nell'Asia centrale o nella regione di Altai. Anche l'aplogruppo Q si originò nell'Asia centrale, migrò verso est e raggiunse l'America del nord attraverso lo stretto di Bering. L'aplogruppo K è caratterizzato dai seguenti aplotipi e dalle seguenti mutazioni che li caratterizzano:
- K M9
- L M11, M20
- M P256
- NOP rs2033003
- NO M214 35-40.000 anni
- N LLY22g
- O M175
- O3 M122
- P M45
- Q M242
- Q3 M3
- R M207, M306
- Q M242
- NO M214 35-40.000 anni
- S M230
- T M70
Aplogruppo R
Tutti gli aplotipi afferenti all'aplogruppo R condividono le mutazioni M207 (UTY2), M306 (S1), S4, S8, S9 e possono essere suddivisi in due principali linee evolutive: R1a e R1b.
La R1a potrebbe essersi originata nelle steppe euroasiatiche a nord del Mar Caspio e del Mar Nero. È associato alla cultura kurgan, nota per la domesticazione del cavallo (circa 5000 anni fa). Questa linea è attualmente presente in Asia centrale e occidentale, India, e nelle popolazioni slave dell'Europa orientale.
La linea R1b è la più comune nelle popolazioni europee. Nell'Irlanda occidentale raggiunge una frequenza prossima al 100%. Si è originata prima della fine dell'ultima glaciazione e si è concentrata nei rifugi del sud-Europa per poi riespandere verso nord con il progressivo mitigarsi del clima a partire da 14.000 anni fa.
Note
- ↑ 1,0 1,1 Elizabeth Wood et al. 2005 Contrasting patterns of Y chromosome and mtDNA variation in Africa: evidence for sex-biased demographic processes European Journal of Human Genetics 13, 867–876. doi:10.1038/sj.ejhg.5201408
- ↑ Michael E. Wealea et al. 2003. Rare deep-rooting Y chromosome lineages in humans: Lessons for Phylogeography. Genetics 165 (1): 229–34
- ↑ Shi H, Zhong H, Peng Y, et al. (2008). "Y chromosome evidence of earliest modern human settlement in East Asia and multiple origins of Tibetan and Japanese populations". BMC Biol. 6: 45. doi:10.1186/1741-7007-6-45. PMID 18959782. PMC 2605740. http://www.biomedcentral.com/1741-7007/6/45.
- ↑ Tatiana Karafet et al 2008, New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree
- ↑ Y-DNA Haplogroup C and its Subclades - 2010
- ↑ Georgi Hudjashov, Toomas Kivisild, Peter A. Underhill et al., "Revealing the prehistoric settlement of Australia by Y chromosome and mtDNA analysis," PNAS, vol. 104, no. 21, pp. 8726–8730 (May 22, 2007)
- ↑ Laura Scheinfeldt et al 2006, "Unexpected NRY Chromosome Variation in Northern Island Melanesia," Molecular Biology and Evolution 23(8):1628–1641. doi:10.1093/molbev/msl028
- ↑ Cordaux, Richard et al 2004, Independent Origins of Indian Caste and Tribal Paternal Lineages
- ↑ Y-DNA Haplogroup G and its Subclades - 2010
- ↑ R. Spencer Wells et al.: "The Eurasian Heartland: A continental perspective on Y-chromosome diversity." Proceedings of the National Academy of Sciences of the United States of America v.98(18); Aug 28, 2001
- ↑ Rootsi S, Magri C, Kivisild T, et al. (July 2004). "Phylogeography of Y-chromosome haplogroup I reveals distinct domains of prehistoric gene flow in europe". Am. J. Hum. Genet. 75 (1): 128–37. doi:10.1086/422196. PMID 15162323. PMC 1181996. http://linkinghub.elsevier.com/retrieve/pii/S0002-9297(07)62002-3.
- ↑ Semino O, Magri C, Benuzzi G, et al. (May 2004). "Origin, diffusion, and differentiation of Y-chromosome haplogroups E and J: inferences on the neolithization of Europe and later migratory events in the Mediterranean area". Am. J. Hum. Genet. 74 (5): 1023–34. doi:10.1086/386295. PMID 15069642. PMC 1181965. http://linkinghub.elsevier.com/retrieve/pii/S0002-9297(07)64366-3.
- ↑ Wells RS, Yuldasheva N, Ruzibakiev R, et al. (August 2001). "The Eurasian heartland: A continental perspective on Y-chromosome diversity". Proc. Natl. Acad. Sci. U.S.A. 98 (18): 10244–9. doi:10.1073/pnas.171305098. PMID 11526236. PMC 56946. http://www.pnas.org/cgi/pmidlookup?view=long&pmid=11526236.
- ↑ 14,0 14,1 Kayser M, Brauer S, Weiss G, Schiefenho¨vel W, Underhill P, Shen P, Oefner P, Tommaseo-Ponzetta M, Stoneking (2003) Reduced Y-Chromosome, but Not Mitochondrial DNA, Diversity in Human Populations from West New Guinea Am J Hum Genet 72:281–302
- ↑ Siiri Rootsi, Lev A Zhivotovsky, Marian Baldovič et al., "A counter-clockwise northern route of the Y-chromosome haplogroup N from Southeast Asia towards Europe," European Journal of Human Genetics (2007) 15, 204–211
- ↑ Map of Y Haplogroups
- ↑ Maria-Catira Bortolini et al 2003 Y-Chromosome Evidence for Differing Ancient Demographic Histories in the Americas
- ↑ Peter A Underhill et al 2009, Separating the post-Glacial coancestry of European and Asian Y chromosomes within haplogroup R1a
- ↑ Y-Chromosome Biallelic Haplogroups
- ↑ Manoukian, Jean-Grégoire (2006). A Synthesis of Haplogroup R2
- ↑ Karafet, Tatiana et al. (2008), New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree, Genome Research, Template:Doi
- ↑ Kivisild et al 2003, The Genetic Heritage of the Earliest Settlers Persists Both in Indian Tribal and Caste Populations
Bibliografia
- http://ycc.biosci.arizona.edu/ Y Chromosome Consortium
- 2005 Y-chromosome Phylogenetic Tree, from FamilyTreeDNA.com
- A Nomenclature system for the Tree of Human Y-Chromosomal Haplogroups, Genome.org
Voci correlate
- Adamo cromosomico
- Aplogruppi mitocondriali umani
- Ipotesi genetiche sul popolamento dell'Europa
- Genetica dei Sardi
- Teoria della catastrofe di Toba